1.Mismatch Repair (MMR) Mekanizması
MMR, DNA replikasyon hatalarını düzelten bir mekanizmadır. Replikasyon sırasında yanlış eşleşen bazları (örneğin, A ile G) tespit eder ve düzeltilmesini sağlar.
Adımlar ve Moleküler Mekanizma
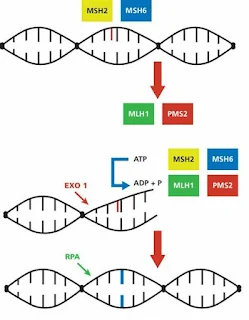
1. Tanıma ve İşaretleme: MutS homodimeri veya heterodimeri (MutSα/Mutsβ) yanlış eşleşmiş bazları tanır ve onarımın başlatılmasını sağlar. Yanlış eşleşme tanındığında, MutS bu bölgeye bağlanır ve konformasyonel değişikliğe uğrar.
2. MMR Kompleksinin Oluşumu: MutLα (MLH1-PMS2 heterodimeri) MutS'ye bağlanarak MutH'yi aktive eder. MutH, yeni sentezlenen DNA'nın metillenmemiş zincirini tanır ve bu zinciri keser.
3. Eksonükleaz Aktivitesi: Kesilen bölgeden itibaren eksonükleaz (Exo1) hasarlı DNA bölgesini uzaklaştırır.
4. DNA Sentezi ve Ligasyonu: DNA polimeraz δ eksik nükleotidleri doğru şekilde ekler. DNA ligaz bu yeni sentezlenen DNA'yı orijinal zincire bağlayarak onarım sürecini tamamlar.
Özel Durumlar:
- E.coli: MutH metillenmemiş zinciri tanır ve keser.
- Eukaryotlar: Hata tanıması için metilasyon durumu gerekli değildir. İnsanlarda MutH homologu bulunmamaktadır.
2. SOS Yanıtı Mekanizması
SOS yanıtı, bakterilerde DNA hasarının algılanmasına ve onarım genlerinin ekspresyonuna yanıt olarak ortaya çıkan acil durumdur.
Adımlar ve Moleküler Mekanizma:
1. Hasarın Algılanması: DNA hasarı meydana geldiğinde, tek zincirli DNA (ssDNA) ortaya çıkar ve RecA proteini bu ssDNA'ya bağlanır.2. RecA Aktivasyonu: RecA, LexA repressörünün oto-katalitik yıkımını indükler. LexA baskılayıcı etkisini kaybettiğinde, SOS genlerinin ekspresyonunu serbest bırakır.3. Onarım Genlerinin Ekspresyonu: DNA hasarını onarmak için UVRABC eksizyon onarım kompleksi ve hata eğilimli polimerazlar (Pol IV, Pol V) gibi SOS genleri eksprese edilir.4. Hata Eğilimli DNA Sentezi: Hata eğilimli DNA polimerazlar, hasarlı bölgeyi geçici olarak onarır ancak genellikle mutasyonlara neden olur.
3. Non-Homologous End Joining (NHEJ) Mekanizması
NHEJ, çift sarmallı DNA kırıklarının homolog bir şablon kullanmadan onarılmasını sağlayan bir mekanizmadır.
Adımlar ve Moleküler Mekanizma:
1. Kırık Uçların Tanınması: Ku70/Ku80 heterodimeri kırık DNA uçlarına bağlanarak DNA-PKcs'yi (DNA-Dependent Protein Kinase Catalytic Subunit) toplayan bir platform oluşturur.
2. DNA Uçlarının İşlenmesi: Artemis endonükleaz, DNA-PKcs ile birlikte çalışarak kırık uçları işlemden geçirir ve mikrosatellit ekleme/silmelerine neden olabilir.
3. DNA Ligasyonu: DNA ligaz IV, XRCC4 ve XLF/Cernunnos ile etkileşime girerek DNA uçlarını doğrudan birleştirir.
Özel Durumlar:
- NHEJ genellikle genetik materyalin küçük kayıplarına neden olur, çünkü kırık uçların yeniden birleştirilmesi sırasında ekleme veya silme hataları oluşabilir.
- Hücre döngüsünün tüm aşamalarında aktiftir, ancak özellikle G1 fazında tercih edilen bir mekanizmadır.
4. Homolog Rekombinasyon (HR) Mekanizması
HR, homolog bir DNA dizisini şablon olarak kullanarak çift sarmallı kırıkları doğru bir şekilde onarır.
Adımlar ve Moleküler Mekanizma:
1. Rekombinasyonun Başlatılması: Kırık uçlar, MRE11-RAD50-NBS1 (MRN) kompleksi tarafından işlemden geçirilir ve rezeke edilir. Bu işlem sırasında tek zincirli DNA (ssDNA) oluşturulur.
2. RAD51 Filamenti Oluşumu: RPA proteini ssDNA'ya bağlanır ve ardından RAD51, BCRA2 yardımıyla bu ssDNA'ya bağlanarak bir nükleoprotein filament oluşturur.
3. Homolog Arama ve İnvazyon: RAD51 filamentleri, homolog kromatidi arar ve bu şablon ile eşleşme sağlar. Eşleşme olduğunda, kırık uçlar homolog şablona invaze olur ve çapraz yapılar (holliday bağlantıları) oluşur.
4. DNA Sentezi ve Holliday Bağlantısının Çözülmesi: DNA polimeraz, homolog şablonu kullanarak eksik DNA'yı sentezler. Holliday bağlantıları çözülür ve onarım tamamlanır.
Özel Durumlar:
- HR, hücre döngüsünün S ve G2 fazlarında daha aktiftir, çünkü bu fazlarda kardeş kromatidler mevcut olduğundan yüksek doğruluk sağlar.
- BRCA1/2 proteinleri, HR mekanizmasında kritik roller oynar ve bu proteinlerdeki mutasyonlar, onarım hatalarına ve kanser riskine yol açabilir.
5. Fotoreaktivasyon Onarımı Mekanizması
Fotoreaktivasyon, UV ışınları nedeniyle oluşan timin dimerlerini onarır ve genellikle birçok bakteri, mantar, bitki ve bazı hayvan türlerinde bulunur.
Adımlar ve Moleküler Mekanizma:
1. Fotoliyaz Aktivasyonu: Fotoliyaz enzimi, timin dimerlerini tanır ve DNA'ya bağlanır. Görünür ışığı emerek aktive olur ve içerdiği kofaktörler (FADH, MTHF) aracılığıyla enerji transferi yapar.2. Dimerlerin Parçalanması: Fotoliyaz, görünür ışığın enerjisini kullanarak timin dimerlerinin bağını kırar. Bu işlem, dimerlerinin bağını kırar. Bu işlem, dimerleri normal timin bazlarına ayırır ve DNA'nın özgün yapısını geri kazandırır.
Özel Durumlar
- İnsanlarda bu mekanizma bulunmaz; bunun yerine, nükleotid eksizyon onarımı (NER) UV kaynaklı hasarı onarımda görev alır.
- Fotoreaktivasyon, doğrudan onarım mekanizması olduğu için mutasyon oluşturma riski yoktur.
Kaynaklar
- Modrich, P. (2006). Mechanisms in eukaryotic mismatch repair. Journal of Biological Chemistry, 281(41), 30305-30309.
- Friedberg, E. C., et al. (2006). DNA Repair and Mutagenesis. ASM Press.
- Jasin, M., & Rothstein, R. (2013). Repair of Strand Breaks by Homologous Recombination. Cold Spring Harbor Perspectives in Biology, 5(11), a012740.
- Lindahl, T., & Barnes, D. E. (2000). Repair of Endogenous DNA Damage. Cold Spring Harbor Symposia on Quantitative Biology, 65, 127-133.
- Walker, G. C., et al. (1980). The SOS regulatory system of Escherichia coli. Cell, 24(1), 249-254.


.jpeg)
.jpeg)




0 Yorumlar