Yeni Nesil Dizileme
DNA dizileme, bir organizmanın genetik materyalini oluşturan nükleotidlerin sırasını belirlemek için kullanılan bir yöntemdir. DNA, dört farklı nükleotid içerir: adenin (A), timin (T), sitozin (C) ve guanin (G). DNA dizileme teknolojileri, bu bazların hangi sırayla dizildiğini tespit ederek genetik bilgiyi ortaya çıkarır(Dönmez, D., Şimşek, Ö., ve Aka Kaçar, Y., 2015).
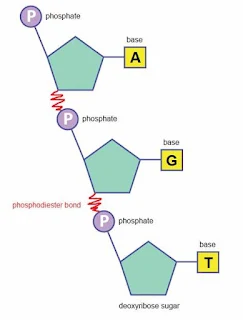 |
| DNA'daki fosfodiester bağları |
Emülsiyon PCR (ePCR) Nedir?
Emülsiyon PCR (ePCR), geleneksel PCR'ın bir varyantı olup, yeni nesil dizileme teknolojilerinde (NGS) sıkça kullanılan bir DNA amplifikasyon yöntemidir. Bu teknik, DNA moleküllerinin amplifikasyonunu (çoğaltılmasını) su ve yağ fazları arasında oluşturulmuş bir emülsiyon ortamında gerçekleştirilir. Emülsiyon PCR, DNA moleküllerinin su damlacıkları içinde izole edilmesi ve her bir damlacığın kendi içinde bağımsız bir PCR reaksiyonu gerçekleştirmesi prensibine dayanır. Bu yöntem özellikle çoklu DNA dizileme (multiplexing) ve yüksek verim gerektiren çalışmalarda tercih edilir.
Emülsiyon PCR'ın Temel Prensibi
1.Emülsiyon Hazırlığı:
- ePCR'de DNA şablonu, yağ-su emülsiyonu içinde dağıtılır. Su fazı içerisinde her bir DNA molekülü, bir damlacıkta izole edilir.
- Her damlacık, bağımsız bir reaksiyonu tüpü gibi davranır ve bu ortamda tek bir DNA molekülü çoğaltılır. Bu sayede her damlacıkta aynı şablonun kopyaları üretir.
2. Amplifikasyon Reaksiyonu:
- Damla içindeki DNA, normal PCR reaksiyonlarına benzer bir süreçle çoğaltılır. Her damlacık, DNA şablonu, primerler, DNA polimeraz ve dNTP'leri içerir.
- Bu bağımsız PCR reaksiyonları sayesinde her damlacıkta, o spesifik DNA molekülüne ait kopyalar elde edilir. Bu yöntem, aynı anda çok sayıda DNA molekülünün çoğaltılmasını sağlar.
3. Su-Damlacık İzolasyonu:
- Her damlacık, farklı DNA moleküllerini birbirinden izole eder ve kontaminasyonu önler. Böylece her damlacıkta çoğaltılan DNA molekülleri, birbirinden bağımsız olur ve farklı DNA şablonları karışmaz.
4. Sonraki Adım:
- Amplifikasyon tamamlandıktan sonra emülsiyon ve çoğaltılan DNA molekülleri sonraki dizileme adımlarına (örn. NGS platformlarına) hazır hale getirilir.
Emülsiyon PCR'nın Avantajları
1. Yüksek Verim:
- ePCR, aynı anda milyonlarca DNA molekülünü amplifiye etme kapasitesine sahiptir. Bu da büyük veri setlerinin analiz edilmesi gereken durumlarda büyük avantaj sağlar.
2. Tek Molekül Hassasiyeti:
- Her damlacıkta tek bir DNA molekülü izole edildiği için, tek bir molekül seviyesinde hassasiyetle amplifikasyon yapılabilir. Bu, az miktarda örnekle çalışırken önemli bir avantaj sağlar.
3. Kontaminasyonun Azaltılması:
- Su damlacıklarının birbirinden izole olması, farklı DNA moleküllerinin karışmasını ve kontaminasyonu önler. Bu, ePCR’nin çoklu DNA dizileme (multiplexing) işlemlerinde kullanılmasına olanak tanır.
4. Yüksek Hassasiyeti:
- Küçük DNA miktarlarını hassas bir şekilde çoğaltabilir, bu da nadir bulunan DNA varyantlarının tespit edilmesini sağlar.
5. Yeni Nesil Dizilime ile Uyum:
- ePCR, özellikle yeni nesil dizileme (NGS) platformlarında yaygın olarak kullanılan bir tekniktir. Örneğin, 454 Pirosekanslama ve Ion Torrent dizileme platformları, DNA amplifikasyonu için emülsiyon PCR'yi kullanır.
Emülsiyon PCR'ın Kullanım Alanları
1. Yeni Nesil Dizileme (NGS):
- ePCR, 454 Pirosekanslama, Ion Torrent ve SOLiD gibi dizileme platformlarında DNA şablonlarının çoğaltılmasında kullanılır. Bu platformlarda çoğaltılan DNA dizileri, daha sonra yüksek hassasiyetle dizilenir.
2. Klonlama ve Genom Analizi:
- Emülsiyon PCR, klonlama işlemlerinde ve büyük ölçekli genom analizlerinde kullanılır. Özellikle metagenomik çalışmalar ve mikroorganizma analizlerinde yaygın olarak tercih edilir.
3. Mutasyon Tespiti:
- Genetik varyantları ve mutasyonları tespit etmek için kullanılan ePCR, az miktarda DNA örneğiyle bile yüksek doğrulukla analiz yapılmasına olanak tanır.
4. Hedeflenmiş Dizileme:
- Özellikle belirli gen bölgelerinin amplifikasyonu ve dizilenmesi gereken durumlarda kullanılır. Bu teknik, hedeflenmiş gen panelleri ve kanser genetiği çalışmalarında da önemli bir rol oynar.
Emülsiyon PCR'ın Dezavantajları
1. Karmaşıklık:
- ePCR, standart PCR’ye göre daha karmaşıktır ve özel laboratuvar ekipmanları gerektirir. Emülsiyon hazırlama ve kırma işlemleri ekstra adımlar içerir.
2. Optimizasyon Gereksinimi:
- Reaksiyon koşullarının doğru bir şekilde optimize edilmesi gerekir. Damla boyutu, reaksiyon bileşenleri ve emülsiyon stabilitesi gibi faktörler sonuçları etkileyebilir.
3. Damlacıkların Eşit Olmaması:
- Emülsiyon oluşturma sırasında her damlacık eşit miktarda DNA içermeyebilir. Bu da bazı damlacıklarda amplifikasyonun yetersiz olmasına neden olabilir.
Birincil Nesil Dizileme: Sanger Dizileme
Sanger Dizileme, Birinci Nesil Dizileme teknolojilerinin en bilinen ve en yaygın kullanılan yöntemlerinden biridir. 1977'de Frederick Sanger tarafından geliştirilmiş olup, DNA'nın nükleotid dizisinin belirlenmesinde uzun yıllar boyunca altın standart olarak kabul edilmiştir. Bu yöntem, DNA replikasyonu sırasında dizilenecek DNA’ya özgü bir şekilde belirlenmiş nükleotidlerin zincir sonlandırılması prensibine dayanır.
Sanger Dizileme Yönteminin Prensibi
Sanger dizileme, DNA polimeraz enziminin doğal replikasyon sürecine dayanır, ancak bu süreçte dizilimi durdurmak için modifiye edilmiş dideoksinükleotidler (ddNTP) kullanılır. Dideoksinükleotidler (ddATP, ddCTP, ddGTP, ddTTP), normal deoksinükleotidlerden (dATP, dCTP, dGTP, dTTP) farklı olarak 3' hidroksil (OH) grubuna sahip değildir. Bu yüzden bir dideoksinükleotid DNA zincirine eklendiğinde, zincirin uzaması durur. Bu özellik, her bir nükleotid eklenmesinden sonra dizilimin hangi bazda durduğunu tespit etmemize olanak tanır.
Sanger Dizileme Adımları
1. DNA Şablonunun Hazırlanması:
- Dizilenecek DNA örneği önce tek zincir haline getirilir.
- Dizilenecek bölgeye özgü bir primer eklenir. Primer, DNA polimerazın yeni DNA zincirini başlatmasına olanak tanır.
2. Reaksiyon Karışımı:
- Reaksiyon karışımında, şablon DNA ve primerin yanı sıra, dört çeşit normal deoksinükleotid (dNTP) ve düşük miktarda dideoksinükleotidler (ddNTP) bulunur.
- DNA polimeraz enzimi, şablon DNA’yı kullanarak yeni bir DNA zinciri sentezler ve normal dNTP’leri zincire ekler.
3. Dideoksinükleotidlerin Zincir Sonlandırması:
- Ancak, DNA polimeraz, zaman zaman ddNTP'leri de zincire ekler. ddNTP’ler eklenince, zincirin 3' OH grubu olmadığından DNA sentezi sona erer.
- Her baz için farklı floresan renkte işaretlenmiş ddNTP'ler kullanılır (ddATP, ddCTP, ddGTP, ddTTP). Bu sayede hangi bazda zincirin sonlandığı belirlenebilir.
4. Elektroforez:
- Dizileme işlemi sonunda, farklı uzunluktaki DNA parçacıkları elde edilir. Bu parçacıklar, baz sırasına göre farklı uzunluktadır.
- Elde edilen DNA parçaları, kapiler elektroforez ile boyutlarına göre ayrıştırılır. Küçük DNA parçaları daha hızlı hareket ederken, büyük parçalar daha yavaş hareket eder.
- Kapiler tüpte hareket eden DNA parçaları, sonunda bir floresan dedektör ile tespit edilir.
5. Sinyal Tespiti ve Analiz:
- Floresan işaretler, elektroforez sırasında tespit edilir ve hangi bazda zincirin sonlandığı belirlenir.
- Sonuç olarak, elde edilen floresan sinyalleri analiz edilerek DNA dizisi sıralı şekilde belirlenir.
 |
| Sanger Sequancing |
Sanger Dizilemenin Avantajları
- Yüksek Doğruluk: Sanger dizileme, oldukça yüksek doğruluk oranına sahiptir (yaklaşık %99.99 doğruluk). Bu nedenle hala küçük ölçekli dizileme projelerinde veya doğrulama çalışmalarında tercih edilir.
- Uzun Okuma Uzunluğu: Yaklaşık 800-1000 baz çiftine kadar dizilme uzunluğu sunar. Bu da özellikle yeni nesil dizileme yöntemleriyle kıyaslandığında bir avantaj olarak görülür.
- Geniş Kullanım Alanı: Genom, plazmid ve PCR ürünlerinin dizilenmesi, genetik mutasyonların tespiti, doğrulama amaçlı dizilemeler gibi çok sayıda alanda kullanılır.
Sanger Dizilemenin Dezantajları
- Düşük Verim: Aynı anda yalnızca bir DNA molekülü dizilenebilir. Bu, yüksek verimli yeni nesil dizileme teknolojilerine kıyasla önemli bir dezavantajdır.
- Yavaş ve Maliyetli: Sanger dizileme, özellikle büyük genomlar söz konusu olduğunda oldukça yavaş ve maliyetli bir yöntemdir.
- Sınırlı Uygulamalar: Büyük genom projeleri, büyük miktarda veri gerektiren çalışmalarda yetersiz kalır. Bu nedenle, daha karmaşık veya geniş çaplı projelerde yeni nesil dizileme teknolojileri tercih edilmektedir.
Sanger Dizilemenin Uygulama Alanları
1. Küçük Genom Uygulamaları:
- Plazmidler, virüs genomları, bakteriyel genomlar gibi daha küçük genomların dizilenmesi için uygundur.
2. Mutasyon Tespiti ve Doğrulama:
- Yeni nesil dizileme projeleri sırasında keşfedilen genetik varyantlar ve mutasyonların doğrulanması için kullanılabilir.
3. Genetik Hastalıkların Analizi:
- Genetik hastalıklara yol açan mutasyonları belirlemek ve doğrulamak için yaygın bir şekilde kullanılır.
4. Klonlama Sonrası Analizi:
- Bir genin klonlanması sonrası doğru dizilimi doğrulamak amacıyla kullanılır.
Yeni Nesil Dizilemenin Tarihçesi
Yeni nesil dizileme teknolojisi 2005 yılında ilk olarak 454 pirodizileme teknolojisinin kullanımı ile ticari olarak erişilebilir duruma gelmiştir. O zamanda beri birkaç farklı dizileme metodu geliştirilmiştir. 454 GS20 pirodizileme (Roche Applied Science), Solexe 1G (Illumina, Inc.), SOLİD (Applied Biosystems), Heliscope (Helicos, Inc.) ve Ion Torrent olmak üzere yeni nesil dizi analiz teknolojilerine ticari olarak ulaşılabilmektedir(Dönmez, D., Şimşek, Ö., Aka Kaçar, Y., 2015).
Yeni Nesil Dizilemenin Avantajları
- Bir çok dizileme reaksiyonu aynı anda ve paralel yapılarak yüksek hacimli ve hızlı sonuç alınmaktadır.
- Embriyonun genetik kodlarının tüm 24 saatten kısa bir sürede ortaya çıkabiliyor. Bunun sonucunda, birbirinden farklı işlemler yapılmasını gerektiren tanı, güvenilir ve düşük maliyetli olarak yapılabilecektir.
- Barkod yöntemi ile DNA parçalarının hangi örneğe ait olduğu kolayca takip edilebilmektedir.
- Hem kalitatif hem kantitatif sonuç verebilen bir yöntemdir. Bu nedenle aynı anda hem mutasyon analizi hem de kromozomların sayısındaki artma ya da azalma tespit edilmektedir.
Yeni Nesil Dizilemenin Dezavantajları
- Günümüzde kullanılan teknikler arasında pahalı olabilmektedir. Aynı zamanda okuma uzunluğunun kısıtlı olması nedeniyle milyonlarca baz içeren genomun tamamını dizileme çalışmaları bu yöntemle oldukça zor olmakta ve uzun sürmektedir.
- Yeni Nesil DNA Dizileme Teknolojisinin başarılı bir şekilde kullanılması için gelişmiş biyoinformatik analiz araçlarına ihtiyaç duyulmaktadır.
- Okuma uzunluğu ve hata oranı konusu hala geliştirilmektedir.
Ligasyon Aracılığıyla ile Dizileme
Ligasyon aracılığıyla dizileme metodunda floresan etiket ile işaretlenmiş olan değişen uzunlukta oligonükleotid problar kullanılır. DNA fragmentleri probların hibridizasyonuna izin veren bilinen kısa bir dizi ile astarlanır. DNA ligaz flowcell'e eklenir. Floresan görüntülüme hangi probun bağlandığını belirlemek için kullanılır. Bu işlem nükleotid dizilerini değerlendirmek için farklı prob setleri kullanılarak tekrarlanır (Egan, S., ve ark. 2012). Ligasyon aracılığıyla dizileme de ABI/SOLİD, Polonator ve Complete Genomics olmak üzere üç metot vardır.
Ligasyon Aracılığıyla Dizileme: ABI/SOLID
SOLID'in açılımı küçük oligonükleotid ligasyon ve tespit sistemidir. Bu teknoloji 2005 yılında George Church tarafından icat edilmiştir. Daha sonra 2008 yılında, şu anda Life Technologies tarafından satın alınan Applied Biosystems tarafından daha da geliştirilmiş ve pazarlanmıştır(Voelkerding, K. V., Dames, S. A., ve Durtschi, J. D., 2009). Sekanslama reaksiyonu 4 geniş adıma ayrılabilir:
- Kütüphane Hazırlama: DNA kütüphanesi hazırlanırken, örnek DNA küçük parçalara (fragmanlara) bölünür. Daha sonra, bu DNA fragmanlarına adaptör dizileri eklenir. Bu adaptörler, dizileme sırasında DNA'yı tutmak ve tanımak için kullanılır.
- Emülsiyon PCR: Hazırlanan DNA kütüphanesi, emülsiyon PCR adı verilen bir teknikle çoğaltılır. Bu süreçte, her bir DNA fragmanı, yağ damlacıkları içinde kaplanarak tek bir DNA molekülüne sahip mikrotaneciklere bağlanır. Her bir yağ damlacığı, kendi başına bir reaksiyon bölgelesi olarak hareket eder. Bu sayede, yüzbinlerce bağımsız reaksiyon aynı anda gerçekleşir(Schuster, S. C., 2008).
- Mikrotaneciklerin Yüzeye Tutunması: PCR sonrasında, DNA fragmanlarıyla kağlanmış mikro tanecikler, cam bir yüzeye veya benzeri bir dizi platformuna tutturulur. Bu yüzey üzerinde bir dizileme reaksiyonu başlatılır.
- Ligasyon Tabanlı Dizileme: Burada, DNA dizilimi kısa oligonükleotid probların hedef DNA'ya bağlanmasıyla belirlenir. Her bir prob, hedef DNA'daki iki bazlık bir bölgeyi tanır ve ligasyon reaksiyonu ile bağlanır. Bu süreçte, iki bazlık (di-baz) problar kullanılır. Probların sonuna farklı floresan işaretleyiciler eklenmiştir ve her bir işaretleyici belirli iki baz kombinasyonunu temsil eder. Bu işlem çok sayıda döngüde tekrarlanarak DNA dizisinin tamamı okunu(Klug, S. W., ve Cummings, W. R., 2000).
.png) |
| ABI/SOLID Sequancing |
Ligasyon Aracılığıyla Dizileme: Polony Dizileme
Polony dizileme (Polymerase Colony Sequancing), diğer dizileme tekniklerinden farklı olarak, jelde çoğaltılmış DNA kolonileri (poloniler) üzerinde dizileme yapılmasına dayanır(Shendure, J., ve ark., 2005).
- Kütüphane Hazırlığı: DNA örneği küçük parçalara (100-200 baz) bölünür. Adaptörler, DNA'nın dizileme sırasında tanınmasını ve çoğaltılmasını sağlayan tanımlayıcı dizilerdir. Aynı zamanda primer bağlanma bölgelerini de içerir. DNA fragmanları, tek moleküler halindeyken poloniler oluşturulması için kullanılacaktır.
- Polony Amplifikasyon: DNA, ince bir jel içerisine yerleştirir. Her bir DNA molekülü jel içinde ayrı bir bölgeye yerleşir. Bu bölgedeki DNA molekülü polimeraz zincir reaksiyonu ile çoğaltılarak polony adı verilen DNA kolonilerini oluşturur. Bu işlem sonucu yüzeye sabitlenmiş binlerce DNA kolonisinin oluşmasını sağlar.
- Dizileme İşlemi: Her polony üzerinde yer alan DNA dizisi, bir dizi kısa oligonükleotid ile dizilenir. Bu oligonükleotidler, floresan etiketli nükleotidlerden oluşur. Ligasyon işlemi, DNA ligaz enzimi adımında, nükleotidlerin bağlanmasıyla birlikte bir floresan sinyali üretilir(Shendure, J., ve ark., 2005).
 |
| Polony Sequancing |
Ligasyon Aracılığıyla Dizileme: Complete Genomics
Büyük ölçekte genom dizileme yapmak için geliştirilmiş, ligasyon tabanlı dizileme tekniklerinden biridir. Bu teknoloji, yüksek verimli düşük maliyetli ve tam genom dizilemesi sağlamak amacıyla tasarlanmıştır.
- Kütüphane Hazırlığı: Dizilenmek istenen genom, kısa fragmanlar haline getirilir. Genellikle 200-400 baz çifti uzunluğundaki bu fragmanlar, dizileme için kullanır. DNA fragmanlarına özel adaptör dizileri eklenir. Bu adaptörler, DNA'nın çoğaltılmasını ve ligasyon adımlarında tanınmasını sağlar.
- Nano-Top Hazırlığı: DNA kütüphanesi, çok sayıda küçük (nano-toplara) bağlanır. Her nano-top, tek bir DNA molekülünün binlerce kopyasını içerir. Nano-toplar üzerinde yerleştirilmiş DNA fragmanları, emülsiyon PCR adı verilen bir teknikte çoğaltılır. Her nano-top aynı DNA fragmanlarının kopyaları oluşur(Deschamps, S., ve Campbell, M. A., 2010).
- Ligasyon Tabanlı Dizileme: DNA dizisine komplementer olan floresan etiketli kısa oligonükleotid, diziyi tanıyarak belirli bir nükleotidi temsil eder. Bağlanan oligonükleotidlerin üzerindeki floresan işaretler, hangi nükleotidin bağlandığı gösterir. Farklı nükleotidler farklı floresan renklerle temsil edilir. Ligasyon adımları tekrarlanarak DNA dizisi okunur. Her bir ligasyon adımında farklı bir baz çifti dizileme işlemi gerçekleştirir.
- Veri Toplama ve Dizilim: Floresan sinyallerinin yüksek çözünürlüklü görüntüleri alınır. Bu veriler, hangi nükleotidlerin bağlandığını ve DNA dizisinin nasıl olduğunu belirler. Büyük ölçekli veri analizi yapılarak genom dizisi yeniden oluşturulur. Compelete genomic, büyük genomların dizilemesin de kapsamlı veri işleme adımlarını içerir. Bu analizler, tam genomun hızlı ve doğru bir şekilde belirlenmesini sağlar.
Sentez Yolu ile Dizileme
Sentez yolu ile dizileme teknolojilerinin temel prensiplerden biridir. Bu yöntem, DNA'nın nükleotid bazlarının dizilimini tespit etmek için, DNA polimeraz enziminin kullanıldığı bir süreçtir. Her bir nüklotidin DNA zincirine eklenmesiyle floresan veya diğer sinyaller algılanır, bu sayede DNA dizisi gerçek zamanlı olarak belirlenir.
Sentez Yolu ile Dizileme: 454 Pirosekanslama
2005 yılında ticari olarak Roche firması tarafından piyasaya sürülmüştür. Pirosekanslama, DNA sentezi sırasında açığa çıkan pirofosfat(Ppi) moleküllerinin ışık sinyali oluşturarak her nükelotidin diziye eklendiğini tespit eden bir yöntemdir(Ansorge, W. S., 2009).
- Kütüphane Hazırlığı: DNA örneği kısa fragmanlara ayrılır. Her DNA fragmanına adaptörler eklenir.
- Emülsiyon PCR: DNA fragmanları, bir yağ emülsiyonu içinde mikro taneciklere bağlanarak çoğaltılır.
- Dizileme Platformuna Bağlanma: DNA ile kaplanmış mikro tanecikler, pikotitre plakası adı verilen bir yüzeyde dizilir.
- Pirosekanslama Reaksiyonu: Pirosekanslamanın temel prensibi, DNA polimeraz enziminin sentezlediği her yeni nükleotidin varlığını pirofosfat (Ppi) salınımı aracılığıyla tespit etmektir. dNTP eklenmesi; dizileme sırasında DNA polimeraz her bir kuyudaki DNA'ya uygun olan nükleotdi ekler ve o sırada bir pirofosfat molekülü serbest bırakılır. ATP ve ışık üretimi; serbest kalan pirofosfat ATP sülfürilaz enzimi tarafından ATP'ye dönüştürülür. Bu ATP, lusiferaz enzimi tarafından kullanılarak bir ışık sinyali üretilir(Morozova, O., ve Marra, M. A., 2008).
Sentez Yolu ile Dizileme: Ion Torrent (Tek Nükleotid Ekleme)
Pirosekanslama benzeri bir teknolojidir. 2010 yılında Life Technologies tarafından tanıtılmıştır ve klasik floresan veya optik algılama yöntemlerinden farklı olarak iyon duyarlılığına dayanır. DNA dizilimi sırasında açığa çıkan hidrojen iyonlarının elektriksel sinyalini tespit ederke diziyi okur(Rothberg, J. M., ve ark., 2011).
- Kütüphane Hazırlığı: DNA örneği küçük parçalara ayrılır ve bu DNA parçalarına dizileme adaptörleri eklenir. Bu adaptörler, DNA'nın amplifikasyonu ve dizilenmesi için gereklidir.
- ePCR: Hazırlanan DNA kütüphanesi ePCR ile çoğaltılır.
- Dizileme Reaksiyonu: Çoğaltılmış iletken DNA parçaları, Ion Torrent yarı iletken çipi üzerindeki kuyucuklara yerleştirilir. DNA polimeraz her doğru nükleotid eklenmesi sırasında bir hidrojen iyonu serbest bırakılır, bu da ortamdaki pH değişikliğine sebep olur(Rothberg, J. M., ve ark., 2011).
- Veri Analizi: Her bir kuyucuktan alınan pH sinyalleri, eklenen nükleotidin kimliğini belirler. Yarı iletken çip, her bir nükleotid eklenmesi sırasında ortaya çıkan sinyali anında algılar ve diziyi hesaplar(Rothberg, J. M., ve ark., 2011).
.png) |
| Ion Torrent Dizileme |
Sentez Yolu ile Dizileme: Pacific Biosciences (Tek Nükleotid Ekleme)
Bu dizileme yöntemi, özellikle Single Molecule Real-Time (SMRT) teknolojisi ile bilinir. Bu teknoloji, DNA'nın polimeraz aktivitesini gerçek zamanlı olarak izleyerek uzun okuma uzunlukları ve yüksek hassasiyet sağlayan bir yöntemdir. PacBio dizileme, genellikle diğer yeni nesil dizileme teknolojilerine göre daha uzun DNA dizilerini okuyabilmesiyle öne çıkar ve karmaşık genomların analizinde büyük avantaj sağlar(Levene, M. J., 2003).
- DNA Hazırlığı: PacBio, DNA moleküllerini kısa parçalara bölmek yerine, olabildiğince uzun (birkaç kilobazdan megabazlara kadar) DNA moleküllerini kullanır. Uzun okuma yapabilme kapasitesi, düşük yapısal varyantları ve tekrarlayan bölgeleri okuma da önemli avantaj sağlar. DNA parçalarına iki uçlu adaptör dizileri eklenir ve bir dairesel yapı oluşturulur. Bu yapı, "SMRTbell kütüphanesi" olarak adlandırılır ve dizileme işlemi sırasında polimerazın DNA'yı dairesel olarak tekrar tekrar okuyabilmesini sağlar. Böylece her DNA dizisi birden fazla kez okunabilir(Eid, J., 2009).
- SMRT Hücresi ve Zero Mode Waveguide (ZMW): ZMW'ler, nanometre ölçeğinde küçük yapılar olup, tek bir DNA molekülünü ve polimeraz enzimini içerir. Bu yapı, sadece tek bir nükleotid ekleme olayının izlenmesine izin verecek kadar küçük olduğundan, DNA dizilme sürecini gerçek zamanlı olarak gözlemlemek mümkün olur. ZMW'nin içindeki reaksiyon alanı çok küçük olduğu için, polimerazın her bir nükleotidi eklerken yaydığı floresan sinyaller dış ortama yayılmadan yüksek çözünürlüklü bir şekilde tespit edilebilir(Levene, M. J., 2003).
- Floresan Etiketli Nükleotidlerin Eklenmesi: Her bir nükleotid (A, T, C ve G) farklı bir floresan renkle işaretlenmiştir. DNA polimeraz, her bir nükleotidi eklerken, floresan etiket kısa bir süre parlayarak hangi nükleotidin eklendiğini gösterir. ZMW'de bulunan yüksek hassasiyetli kamera, her nükleotidin eklenme sürecini gerçek zamanlı olarak izler. Polimerazın her nükleotidi eklenmesi sırasında bir ışık sinyali yayılır, bu sinyal kaydedilir ve daha sonra analiz edilerek DNA dizisi belirlenir. SMRTbell yapı sayesinde, DNA molekülü dairesel olarak tekrar tekrar okunur. Aynı DNA dizisi birden fazla kez okunduğundan, hata oranı önemli ölçüde azalır.
- Veri Toplama ve Analizi: Her bir floresan sinyalinin renk ve süre bilgileri toplanır. Bu bilgiler, hangi nükleotidin dizilendiğini ve polimerazın aktivite hızını belirlemek için kullanılır. Her okuma ham verisi (raw reads), tek bir DNA molekülünden elde edilen dizi bilgisini içerir. Bu ham veriler, aynı DNA dizisinin birden fazla kez okunmasıyla güvenilirlik artırılır. PacBio, bu ham verileri işleyecek biyoinformatik yazılımları sunar. Bu yazılımlar, dizilenmiş verileri analiz eder ve doğru DNA dizisini çıkarır. Uzun okuma kapasitesi sayesinde, tekrar eden diziler, karmaşık yapısal varyantlar ve genetik varyasyonlar daha net bir şekilde analiz edilebilir(Levene, M. J., 2003).
.jpeg) |
| Pacific Biosciences |
Sentez Yolu ile Dizileme: Oxford Nanopore (Tek Nükleotid Ekleme)
Oxford Nanopore dizileme yöntemi, DNA veya RNA dizilerinin doğrudan okunmasına olanak tanıyan yenilikçi bir nanopor teknolojisi üzerine kurulur. Diğer dizileme yöntemlerinden farklı olarak, uzun DNA/RNA zincirlerini bir gözenek (nanopor) aracılığıyla geçirerek her bir bazın elektriksel özelliklerini ölçer ve bu sayede diziyi tespit eder. Nanopor dizileme, gerçek zamanlı okuma, düşük maliyetli ve taşınabilirlik gibi benzersiz avantajlar sunar(Greninger, A. L., ve ark., 2015).
- Kütüphane Hazırlığı: Oxford Nanopore dizileme yöntemi, minimum DNA/RNA hazırlığı getirir. Bu, zincirlerin büyük ölçüde bozulmadan doğal hallerinde dizilenmesine olanak tanır. DNA veya RNA molekülünü adaptörler. Bu adaptörler, DNA veya RNA'nın motil protein ile bağlanarak nanoporlardan geçirilmesini sağlar. Kütüphane hazırlığı sırasında herhangi bir PCR aşaması gerekmez, bu da yöntemi özgün kılan unsurlardan biridir.
- Nanopor Dizileme Süreci: Nanopor dizileme, diğer yöntemlerden farklı olarak tek molekül seviyesinde gerçekleştirilir. Yani her bir DNA veya RNA molekülü, bireysel olarak dizilenir. Bu özellik, düşük miktarda örnekten veri elde edilmesini sağlar. Motil protein, DNA veya RNA molekülünü nanopordan geçirir. Geçiş sırasında her bir bazın elektriksel özellikleri sinyal değişiklikleri oluşturur. Bazların nanopordan geçişi sırasında oluşan elektriksel sinyaller bilgisayara aktarılır ve analiz edilir. Her bir bazın oluşturduğu sinyal, DNA dizisinin belirlenmesinde kullanılır. Nanopor dizilemesi sırasında DNA dizisi gerçek zamanlı olarak elde edilir ve analiz edilir. Kullanıcılar, dizileme tamamlanmadan önce bile verileri izleyebilir ve analiz edebilirler(Greninger, A. L., ve ark., 2015).
- Veri Analizi: Her bir bazın nanopordan geçerken yarattığı sinyal, ham veri olarak toplanır. Bu sinyaller, hangi bazların geçtiğini belirlemek için analiz edilir. Oxford Nanopore dizileme cihazları, hızlı veri işleme kapasitesine sahiptir. Genellikle dizileme devam ederken sonuçlar alınabilir ve veriler anında yorumlanabilir. Oxford Nanopore teknolojisinin ilk versiyonlarında, özellikle tek okumalarda hata oranı yüksekti. Ancak, gelişmiş algoritmalar ve birden fazla okuma yöntemiyle bu hata oranı önemli ölçüde azaltılmıştır.
 |
| Oxford Nanopor |
Sentez Yolu ile Dizileme: Illumina (Döngüsel Geriye Dönüşümlü Sonlandırma)
Yeni nesil DNA dizileme teknolojilerinin en yaygın kullanılanlarından biridir. Yüksek doğrulur, hassasiyet ve verimlilik sunar. Milyonlarca DNA fragmanı aynı anda paralel olarak dizileme kapasitesine sahiptir(Morozova, O., ve Marra, M. A., 2008).
- Kütüphane Hazırlığı: DNA örneği kısa fragmanlara ayrılır. Her bir DNA fragmanına adaptör dizileri eklenir. Bu adaptör dizileri primer bağlanma bölgelerini içerir.
- DNA'nın Flow Cell Yüzeyine Bağlanması: Hazırlanan DNA kütüphanesi, flow cell adı verilen bir yonga üzerine eklenir. Flow cell, yüzeyinde DNA'yı tutmak için komplementer dizilere sahip olan binlerce küçük kuyucuk içerir. DNA fragmanları yüzeyde bulunan adaptörlere komplementer bölgeler aracılığıyla bağlanır(Bentley, D. R., 2006):
- Köprüleme Amplifikasyonu (Bridge Amplification): Flow cell yüzeyinde tutanan DNA fragmanları, yüzeyde sabitlenmiş olan ikincil bir adaptör dizisiyle etkileşime girer. Bu sayede her DNA molekülü, yüzeyde çok sayıda kopyasını oluşturan bir kümeye dönüşür(Hutchison, C. A., 2007).
- Sentez Yoluyla Dizileme: DNA polimeraz enzimi, yüzeyde çoğaltılan her DNA molekülü için yeni bir DNA ipliği sentezler. Dört farklı flüoresan etiketli dNTP eklenir. Her bir baz, kendine özgü bir flüoresan işaretleyici taşır. DNA polimeraz bağlandığında, o bazın flüoresan sinyali oluşur ve kaydedilir. Reversible terminatör sayesinde her nükleotid bağlanmasının ardından zincir uzaması durur(Huntchison, C. A., 2007).
 |
| Illumina Sequancing |
 |
| Yeni Nesil DNA Dizileme Metotlarının Karşılaştırılması |
Yeni Nesil Dizilemenin Çalışma Alanları
- Metagenomik: Herhangi bir çevresel örnekte var olan tüm mikrobiyolojik genomların (microbiome) rastgele dizilenmesi sonucunda ortamın mikrobiyolojik çeşitliliğini ortaya çıkarmak mümkündür. Bu çalışmalar çevresel ortamda hangi mikropların ne miktarda olduğu hakkında da fikir verdiği gibi, daha önce bilinmeyen veya çok az bilinen, sadece kendi doğal ortamında büyüyen ama kültür ortamında büyümeyen bakteriler hakkında da bilgi verir(Gilbert, J. A., ve Dupont, C. L., 2011).
- Bitkiler ve Tarımsal Biyoteknoloji: Bitki genomunun karmaşık yapısı ve büyüklüğü nedeniyle Sanger Dizileme teknolojisi kullanıldığında, maliyetin yüksek olmasının yanında yöntem teknolojik olarak da kısıtlı olduğundan bitki projeleri sadece büyük çaplı dizileme merkezleriyle sınırlı kalmaktadır. Yeni nesil dizileme, uzun ve yüksek doğruluk oranlı okumalarla yüksek veri kalitesi sunmaktadır ve uygun hız ve maliyet nedeniyle bitki çalışmalarında oldukça kullanışlıdır(Weber, A. P., ve ark., 2007).
- Atasal DNA: Yeni nesil dizileme sistemleri ile fosilden, saç, kemik gibi donmuş veya korunmuş örneklerden elde edilen DNA'ların geniş kapsamlı analizini yapmak mümkündür. Dizileme sonucunda, atasal hedef diziler çevresel kontaminasyondan ayırt edilebilir(Wheeler, D. A., ve ark., 2008).
- Transkriptom: Transkriptom dizileme, mRNA transkripsiyon-ekspresyon analizi, yeni genlerin keşfi, henüz genomu tamamıyla tanımlanmamış organizmalarda gen bölgelerinin tanımlanması, tüm gen dizisinin oluşturulması, tek nükleotid polimorfizmlerin (SNP) belirlenmesi, insersiyon, delesyon ve splicing varyantların tespiti, allel spesifik ekspresyonların analizi ve kromozomal yeni düzenlenmelerin belirlenmesini kapsar.
- Small RNA'lar: Küçük RNA'lar; MicroRNA'lar, endojen siRNA'lar ve Piwi-interacting RNA’ların dahil olduğu gruptur. Bu sistem sayesinde bakteri içerisinde klonlanma yapılmadan, yüzlerce hatta binlercesi tanımlanabilir ve miktarı belirlenebilir. Daha önce tanımlanmamış küçük RNA'lar tespit edilebilir ve farklı ekspresyon profili çıkartılabilir veya küçük RNA transkriptomları ile karşılaştırılabilir (Parameswaran, P., ve ark., 2007).
- Metilasyon Analizi: Gen aktivasyonunun epigenetik düzenlenmesi, gelişimsel süreçte son derece önemlidir. Birçok hastalığın metilasyondaki değişikliklerle ilgisi olduğu belirlenmiştir.
Kaynakça
- Dönmez, D., Şimşek, Ö., & Kaçar, Y. A. (2015). Yeni nesil DNA dizileme teknolojileri ve bitkilerde kullanımı. Türk Bilimsel Derlemeler Dergisi, (1), 30-37.
- Türktaş, M. (2011). Floresan Temelli Yeni nesil genetik analiz uygulamaları: DNA dizi analizi, moleküler markörler uygulamaları ve çoklu gen anlatım analizleri uygulamalı eğitimi kitapçığı.
- Klug, S. W., & Cummings, W. R. (2000). Concept of genetics (pp. 745). Prentice Hall.
- Voelkerding, K. V., Dames, S. A., & Durtschi, J. D. (2009). Next-generation sequencing: from basic research to diagnostics. Clinical chemistry, 55(4), 641-658.
- Schuster, S. C. (2008). Next-generation sequencing transforms today's biology. Nature Methods, 5, 16-18.
- Ansorge, W. J. (2009). Next-generation DNA sequencing techniques. New Biotechnology, 25(4), 195-203.
- Morozova, O., & Marra, M. A. (2008). Applications of next-generation sequencing technologies in functional genomics. Genomics, 92, 255-264.
- Rothberg, J. M., Hinz, W., Rearick, T. M., Schultz, J., Mileski, W., Davey, M., ... & Bustillo, J. (2011). An integrated semiconductor device enabling non-optical genome sequencing. Nature, 475(7356), 348-352.
- Bentley, D. R. (2006). Whole-genome re-sequencing. Current Opinion in Genetics & Development, 16, 545-552. Hutchison, C. A. (2007). DNA sequencing: Bench to bedside and beyond. Nucleic Acids Research, 35, 6227-6237.
- Gilbert, J. A., & Dupont, C. L. (2011). Microbial metagenomics: Beyond the genome. Annual Review of Marine Science, 3, 347-371.
- Weber, A. P., Weber, K. L., Carr, K., Wilkerson, C., & Ohlrogge, J. B. (2007). Sampling the Arabidopsis transcriptome with massively parallel pyrosequencing. Plant Physiology, 144, 32-42.
- Wheeler, D. A., Srinivasan, M., Egholm, M., Shen, Y., Chen, L., McGuire, A., & He, W. (2008). The complete genome of an individual by massively parallel DNA sequencing. Nature, 452, 872-877. Parameswaran, P., Jalili, R., Tao, L., Shokralla, S., Gharizadeh, B., Ronaghi, M., & Fire, A. Z. (2007). A pyrosequencing-tailored nucleotide barcode design unveils opportunities for large-scale sample multiplexing. Nucleic Acids Research, 35, e130.
- Shendure, J., Porreca, G. J., Reppas, N. B., Lin, X., McCutcheon, J. P., Rosenbaum, A. M., ... & Church, G. M. (2005). Accurate multiplex polony sequencing of an evolved bacterial genome. Science, 309(5741), 1728-1732.
- Deschamps, S., & Campbell, M. A. (2010). Utilization of next-generation sequencing platforms in plant genomics and genetic variant discovery. Molecular breeding, 25, 553-570.
- Eid, J., Fehr, A., Gray, J., Luong, K., Lyle, J., Otto, G., ... & Turner, S. (2009). Real-time DNA sequencing from single polymerase molecules. Science, 323(5910), 133-138.
- Quail, M. A., Smith, M., Coupland, P., Otto, T. D., Harris, S. R., Connor, T. R., ... & Gu, Y. (2012). A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers. BMC genomics, 13, 1-13.
- Greninger, A. L., Naccache, S. N., Federman, S., Yu, G., Mbala, P., Bres, V., ... & Chiu, C. Y. (2015). Rapid metagenomic identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome medicine, 7, 1-13.
- Bundock, P. C., Eliott, F. G., Ablett, G., Benson, A. D., Casu, R. E., Aitken, K. S., & Henry, R. J. (2009). Targeted single nucleotide polymorphism (SNP) discovery in a highly polyploid plant species using 454 sequencing. Plant biotechnology journal, 7(4), 347-354.
- Azam, S., Thakur, V., Ruperao, P., Shah, T., Balaji, J., Amindala, B., ... & Varshney, R. K. (2012). Coverage‐based consensus calling (CbCC) of short sequence reads and comparison of CbCC results to identify SNPs in chickpea (Cicer arietinum; Fabaceae), a crop species without a reference genome. American journal of botany, 99(2), 186-192.
- Margulies, M. Et al. (2005). Genome sequencing in microfabricated high-density picolitre reactors
- Dressman, D. Et al. (2003). Transforming single DNA molecules into fluorescent magnetic particles for detection and enumeration of genetic variations


.jpeg)




0 Yorumlar